1950年代にDNAポリメラーゼというDNAの合成を触媒する酵素が単離されました。1970年代になると制限酵素とDNAリガーゼという酵素を用いて、DNAを切り貼りできるようになりました。この年代はサンガー法、マクサム・ギルバート法、サザンブロッティング法というDNAの塩基配列の解析方法も発表されました。1980年代にはポリメラーゼ連鎖反応法(PCR法)が開発され、DNAの特定の領域を増幅できるようになりました。このように遺伝子工学が発展していきました。
現在ではPCRはDNAの増幅をリアルタイムで測定できるようになりました。また、遺伝子・DNA解析はサンガー法の次の世代である次世代シークエンサー(NGS)や、マイクロアレイといった新しい技術が登場しています。
遺伝子解析技術
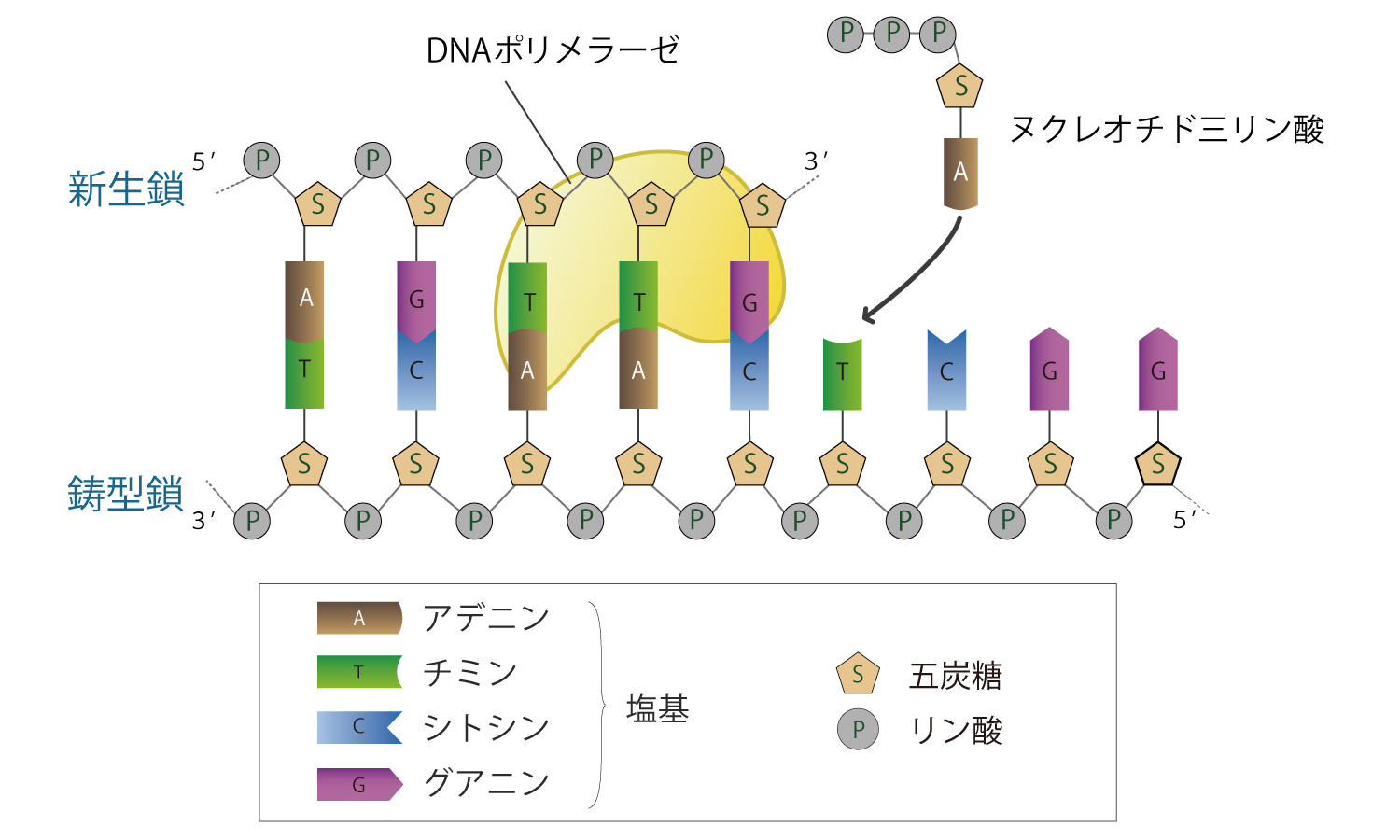
DNA ポリメラーゼ
1本鎖DNAに結合し相補的な塩基をもつヌクレオチドを付加していきます。この酵素により、生体外でDNAを複製できるようになりました。
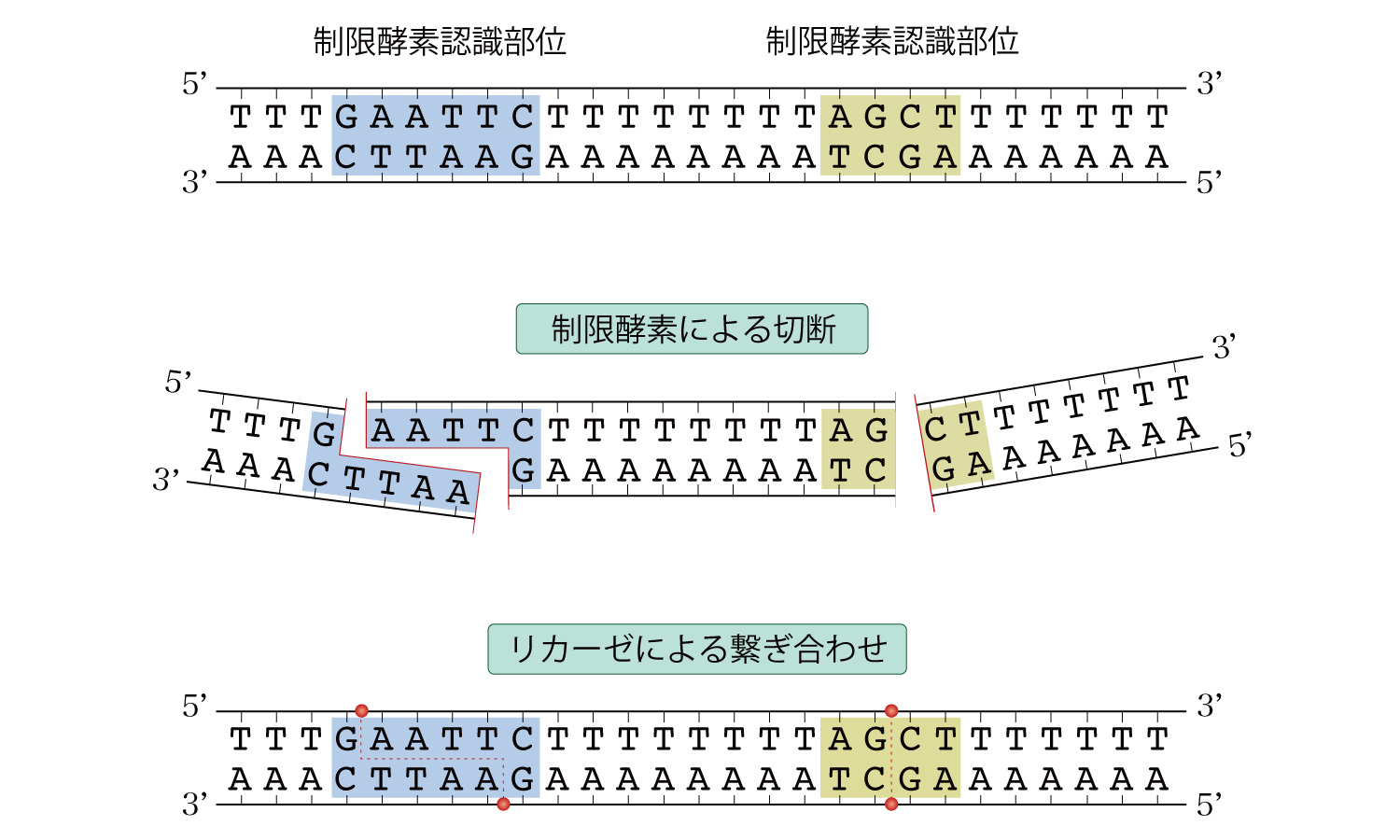
制限酵素・DNAリガーゼ
制限酵素はある特定の塩基配列を認識してDNAを切断します。認識する塩基配列や切断された断面は制限酵素の種類によって異なります。DNAリガーゼは2つのDNA末端を結合する働きをします。この2つの酵素によって、生体外で生物の特定のDNA領域を取り出し、全く別の生物のDNAに組込むことができるようになりました。
サンガー法
サンガー法は現在でも広く用いられているDNA配列決定法です。開発当初は放射性同位体でDNAを標識していましたが、現在では4種類の塩基それぞれに異なった蛍光色素で標識されています。
鋳型となる1本鎖DNA、放射性同位体で標識したプライマー、dNTP(dATP、dGTP、dCTP、dTTP)、DNAポリメラーゼの混合物に、ddNTP(ddATP、ddGTP、ddCTP、ddTTP)をそれぞれ別に加えます。
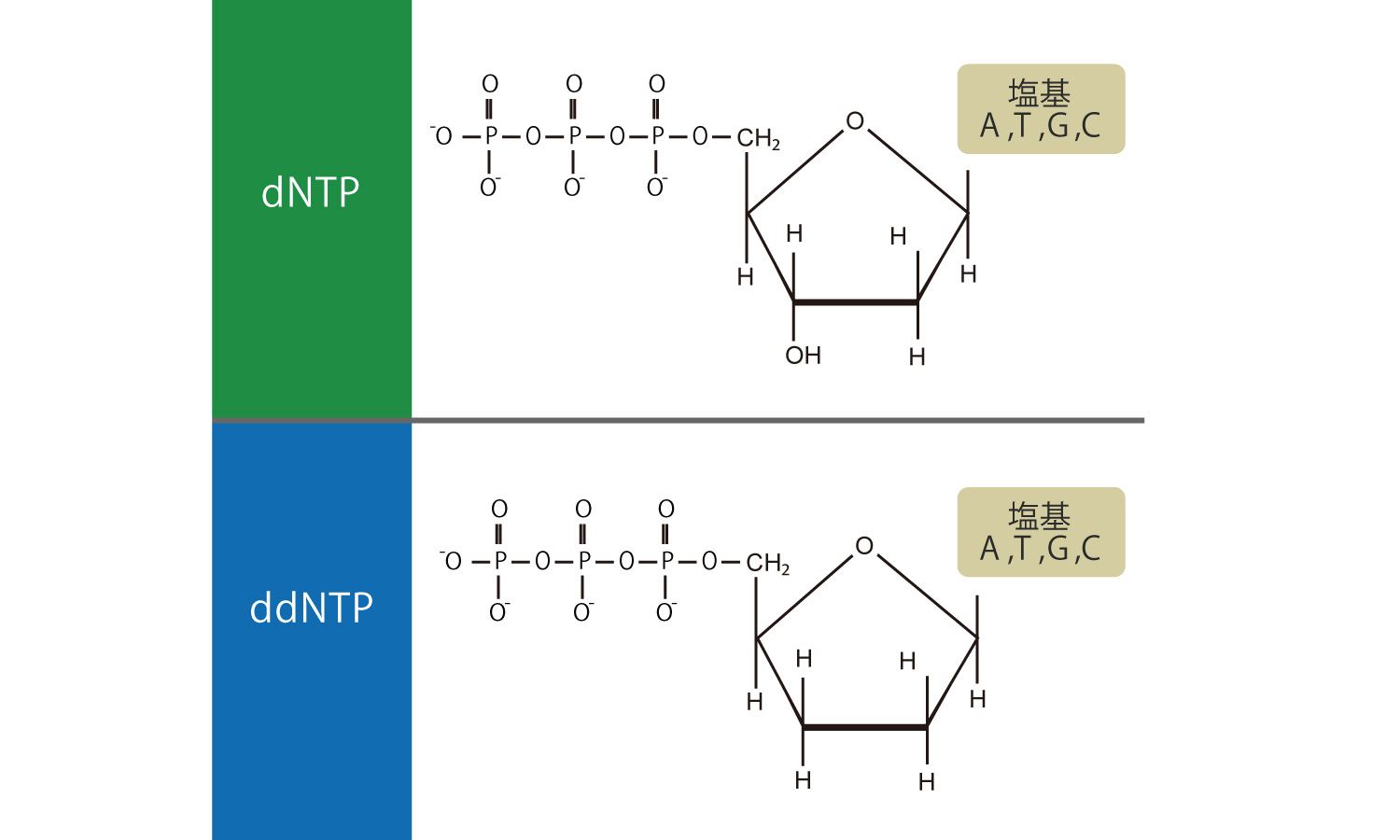
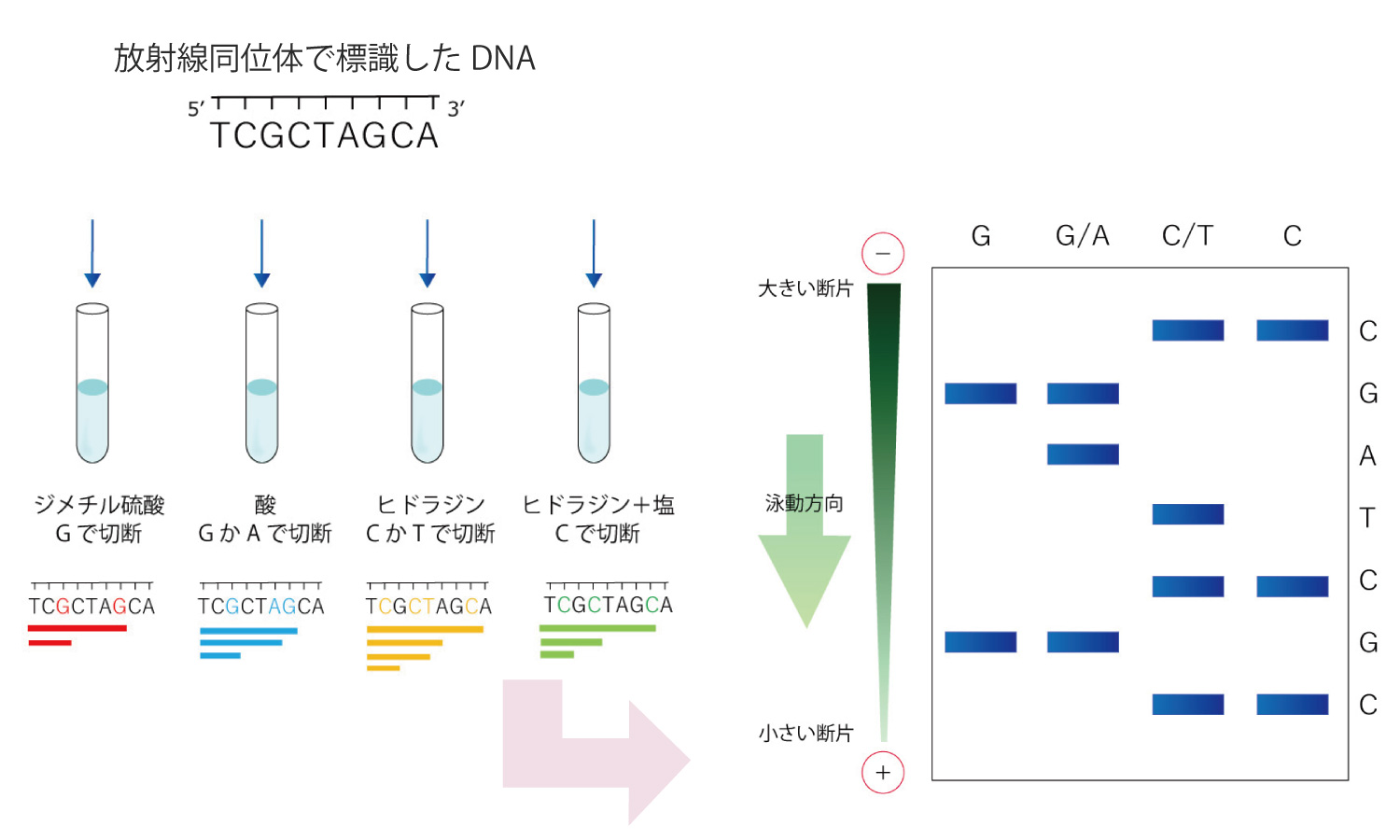
ddNTPが取り込まれるとDNA合成は停止しますが、反応の停止はDNA鎖のランダムな場所で起こるため、長さの異なる様々なDNAが複製されます。この反応物を電気泳動にかけるとDNAの長さによって同じ時間で移動する距離が異なります。これを観察することで、鋳型としたDNAの塩基配列が分かります。
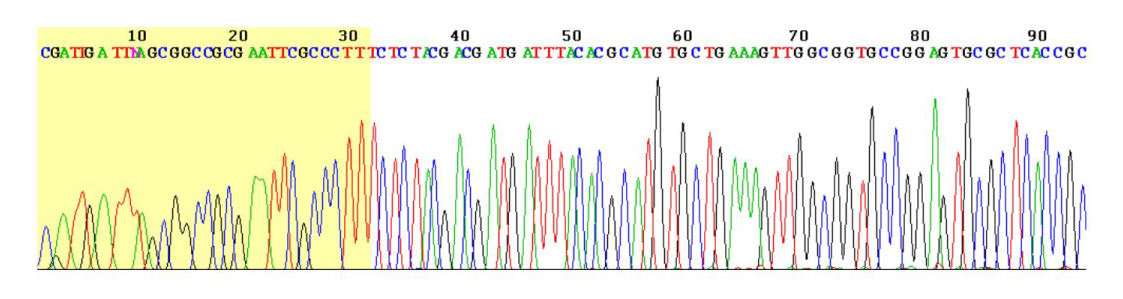
出典元:DNAシークエンシング - Wikipedia
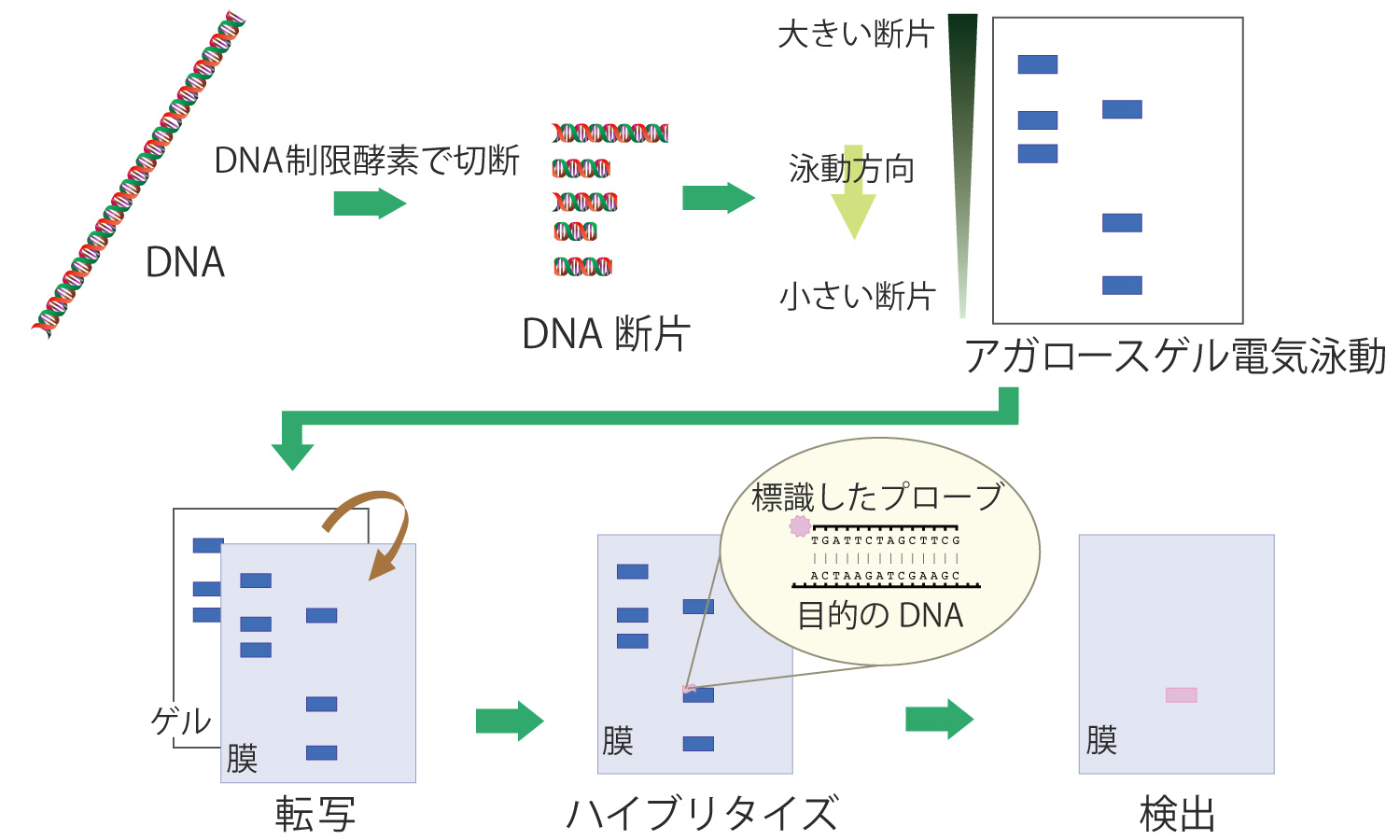
サザンブロッティング
エドウィン・サザンが考案した特定のDNA配列の有無を調べる方法です。DNAを制限酵素で処理し、電気泳動でDNA断片を分離します。DNA断片を1本鎖に変性し膜に転写した後、標識したプローブと結合させ検出します。
なお、サザンブロッティングにちなんで、同様の原理のRNA分析法をノーザンブロッティング、タンパク分析法をウェスタンブロッティングと呼んでいます。
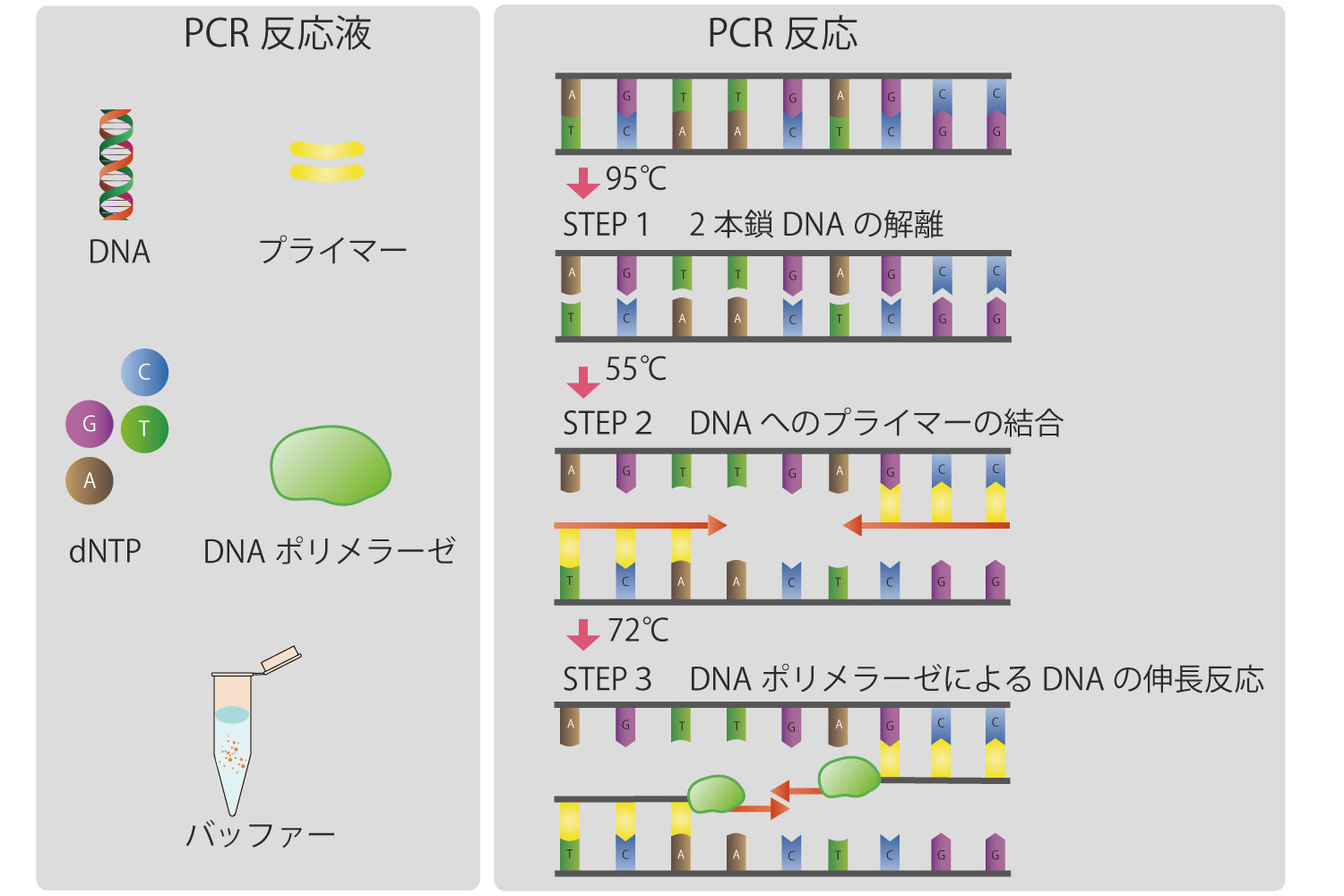
ポリメラーゼ連鎖反応法(PCR法)
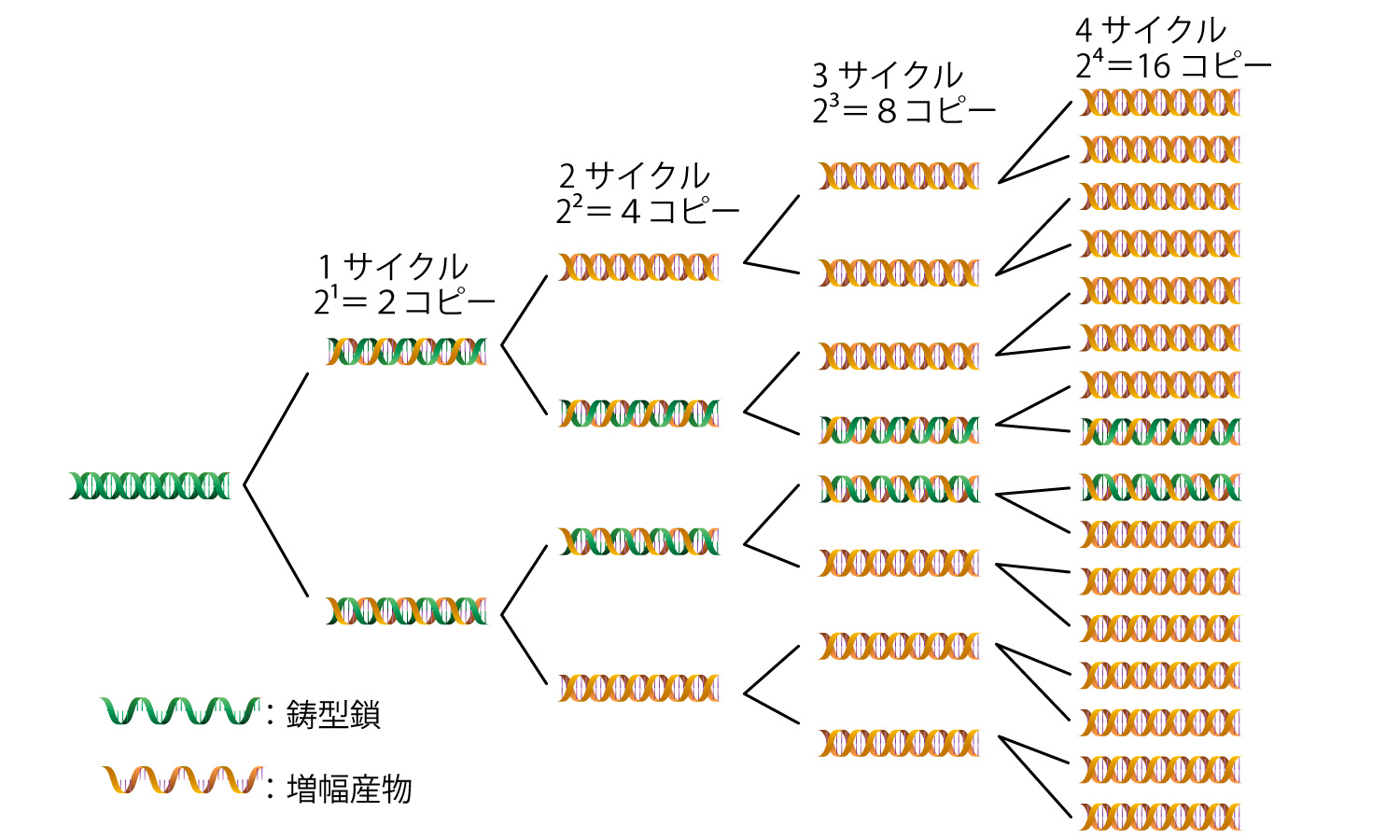
DNAの特定領域を増幅する方法です。 鋳型となる2本鎖DNA、プライマー、dNTP(dATP、dGTP、dCTP、dTTP)、DNAポリメラーゼを含んだ反応液を作製し、次のステップを繰り返します。ステップ1は95℃前後による2本鎖DNAの解離、ステップ2は1本鎖DNAへのプライマーの結合、ステップ3はDNAポリメラーゼによるDNAの伸長反応です。この反応を繰り返すことでDNAの特定領域を増幅することができます。
開発初期は大腸菌から単離したDNAポリメラーゼを使用していましたが、熱で酵素が失活するため、1サイクル毎に酵素を加えていました。1986年に好熱菌(Thermus aquaticus)から見つかった高温でも失活しないDNAポリメラーゼ(Taqポリメラーゼ)が使用されるようになり、操作が簡便になりました。
逆転写ポリメラーゼ連鎖反応(RT-PCR法)
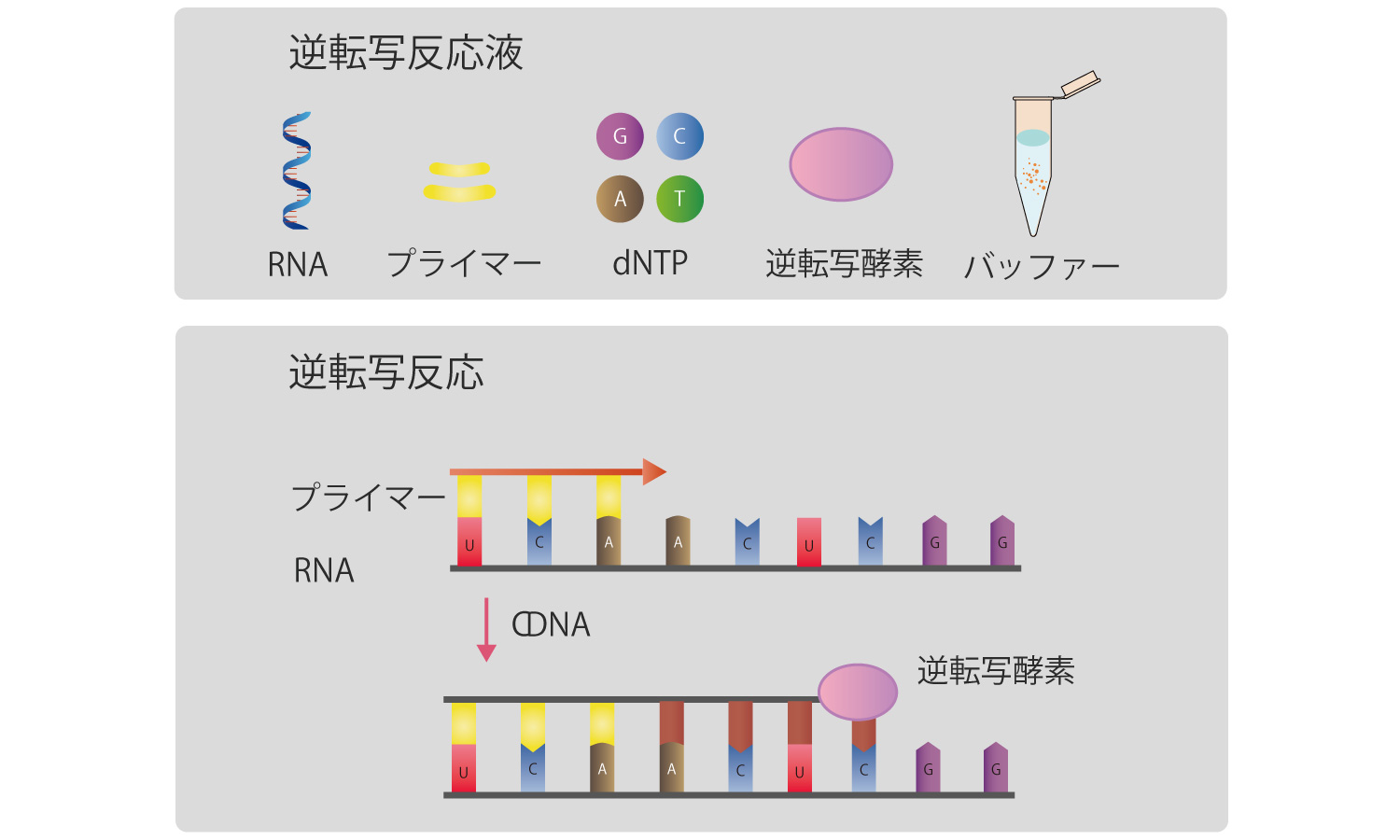
RNAの特定領域を増幅させる方法です。PCR反応はTagポリメラーゼという耐熱性のDNA合成酵素を使用するため、RNAは増幅することができません。そのため、一本鎖RNAを鋳型としてDNA合成を行う逆転写酵素を使用した反応でRNAからDNAを合成します。逆転写反応は、RNA、dNTP(dATP、dGTP、dCTP、dTTP)、逆転写酵素を含んだ反応液を作製し行います。この反応物はcDNA(相補的DNA)とよばれ、次にcDNAに対してPCR反応を実施します。
逆転写反応とPCR反応を1ステップで行う方法と、逆転写反応とPCR反応を別々に行う2ステップの方法があります。2ステップの方法は逆転写とPCRでプライマーを変えることができます。
RT-PCRは、mRNAの検出やRNAウイルスの検出などに用いられています。

リアルタイムPCR、qPCR
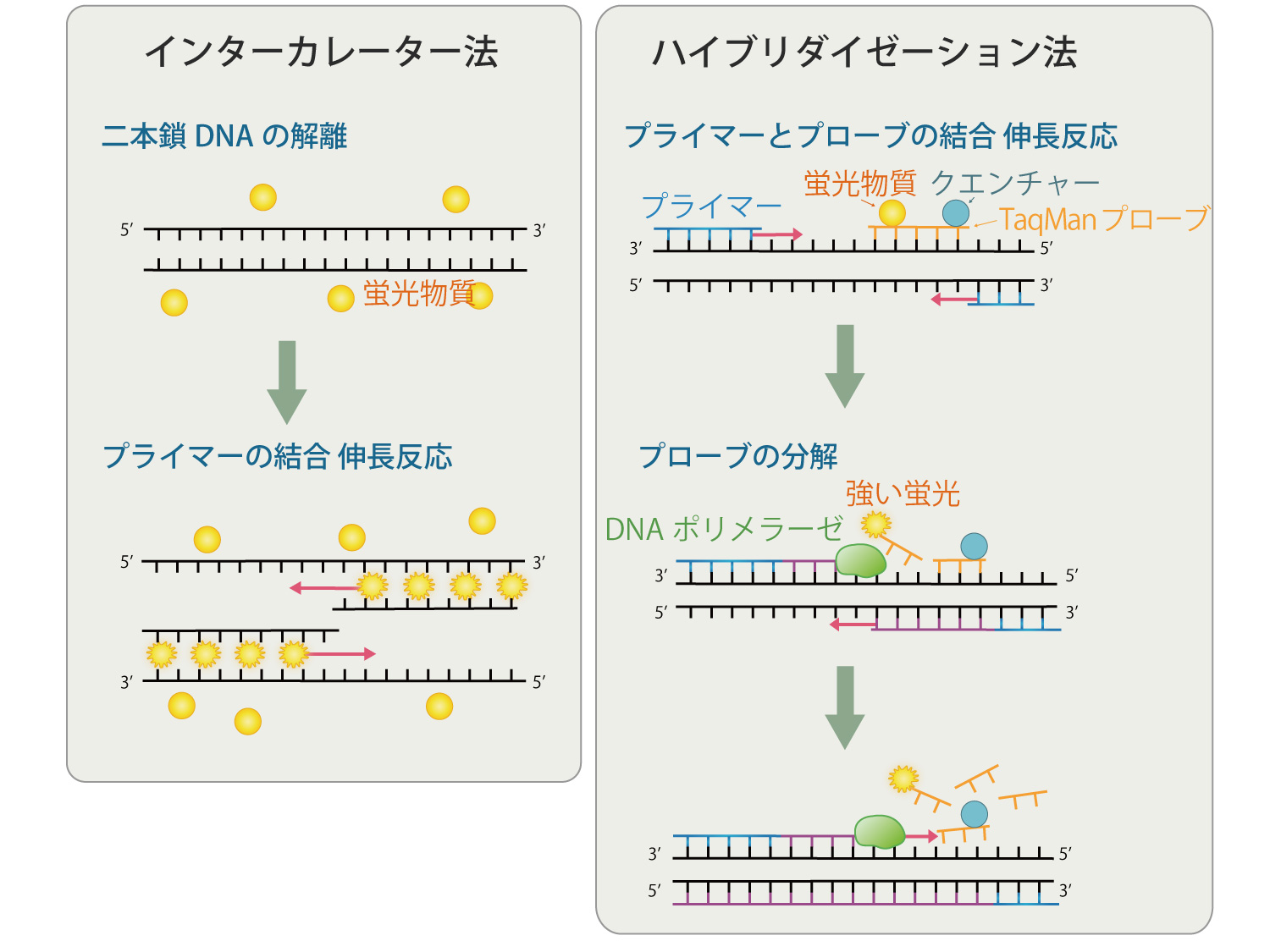
PCRによるDNAポリメラーゼ連鎖反応の増幅数をリアルタイムで測定する方法です。qPCRとも言います。インターカレーターを用いる方法と、プローブを用いる方法があります。
インターカレーター法は、2本鎖DNAに結合すると強い蛍光を示す物質を使用します。蛍光物質であるインカレーターがPCRで複製された2本鎖DNAの間に入り込み強い蛍光を示します。
ハイブリダイゼーション法ではTaqManプローブを使用した例を紹介します。オリゴヌクレオチドに蛍光物質とその発光を抑える働きをするクエンチャーを付加したTaqManプローブを使用します。PCRの過程でTaqManプローブは目的のDNAに結合するのですが、この段階では強い発光を示しません。プライマーからDNAの合成が始まり、TaqManプローブまで伸長するとDNAポリメラーゼがTaqManプローブを切断し、蛍光物質がクエンチャーと離れ強い蛍光を示します。
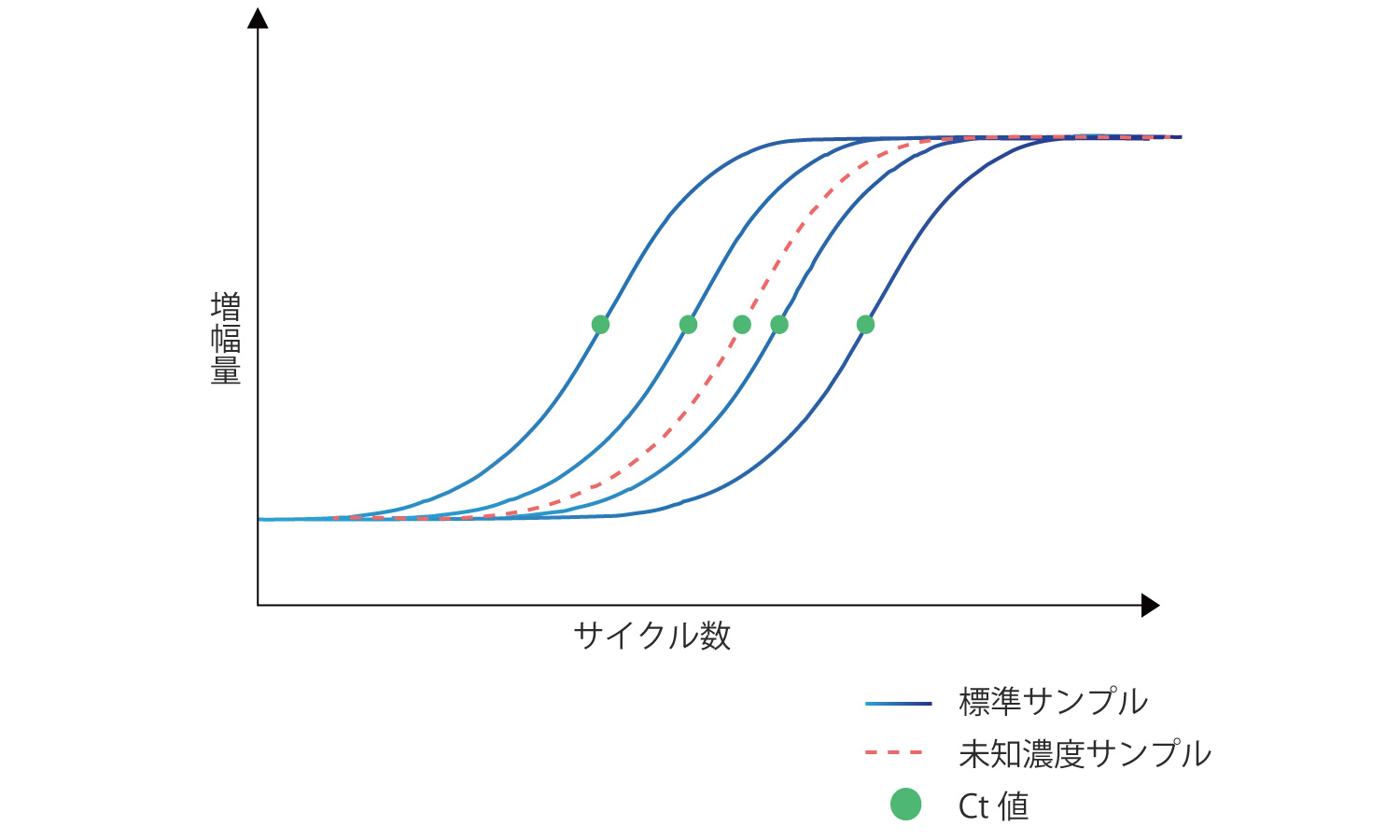
このようにして得られる蛍光強度を測定することでDNAの増幅を測定することができます。
上述のRTーPCRと組み合わせることによって、様々なRNAの定量解析を迅速に行うことが可能となっています。
次世代シークエンサー(NGS)
サンガー法では長さの異なるDNAを合成した後に、分子ふるいで分離しながら塩基を読んでいました。次世代シークエンサーではDNAを合成しながら測定することができるようになりました。 ここでは原理の異なる2つの方法をご紹介します。
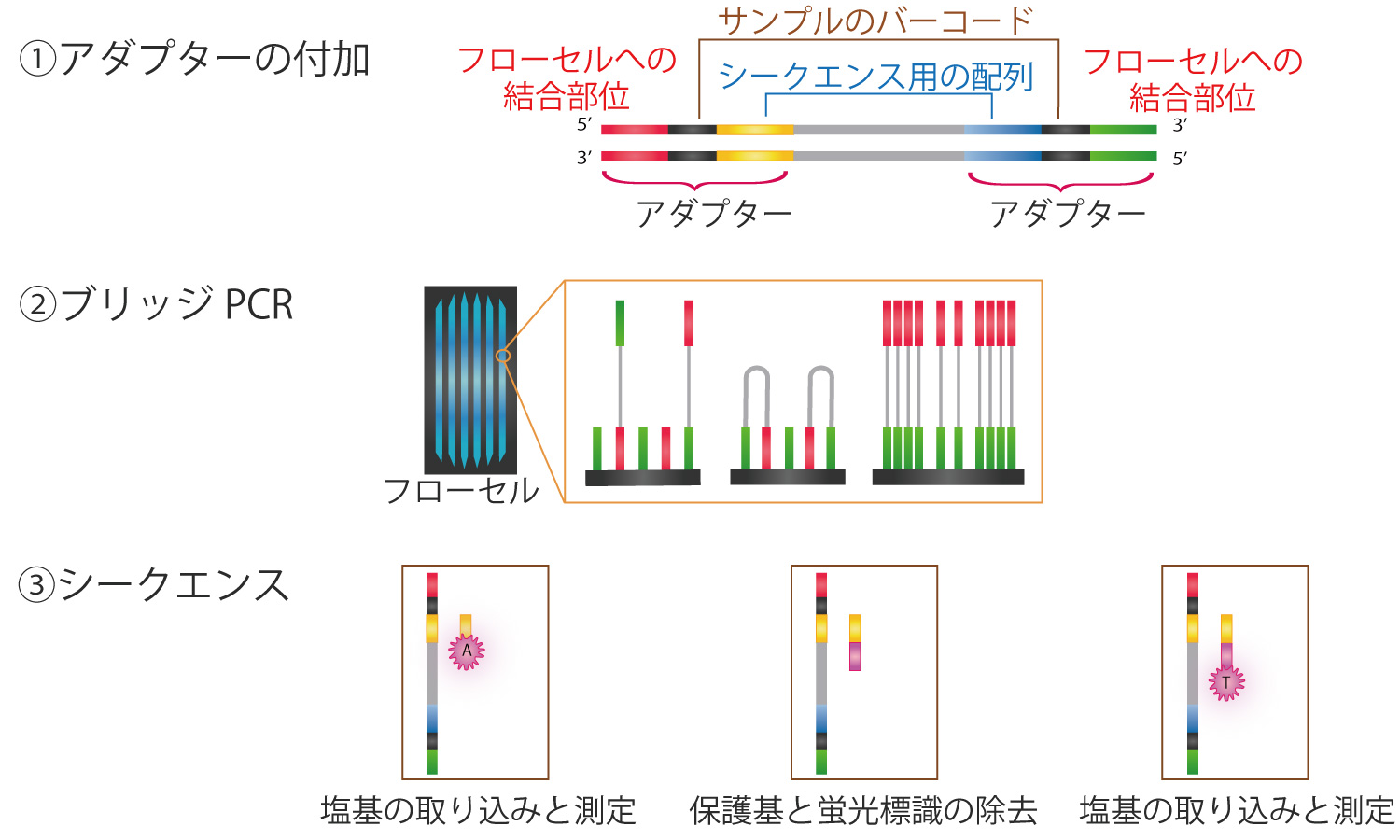
Illumina社のSequencing By Synthesis(SBS) system
ブリッジとPCRとDNA伸長によるシークエンス方法です。
数百bpのDNA断片にサンプルが特定できるバーコードとフローセルへの結合部位、シークエンス用配列を含んだアダプターを付加します。
フローセルはガラスの基盤にアダプターに相補的なオリゴヌクレオチドが付加されています。変性された1本鎖DNAがフローセルのオリゴヌクレオチドに結合し、増幅が開始されます。アダプターの片方の部位が別のオリゴヌクレオチドとブリッジPCRすることでDNAが増幅します。この反応が何回も行われクラスターが形成されます。
シークエンスは1塩基伸長する毎に蛍光を読み取ります。伸長方向を保護基でブロックし、かつ蛍光標識したdNTPが1塩基分取り込まれます。蛍光を測定し、保護基と蛍光標識を除き、次の塩基を取り込みます。
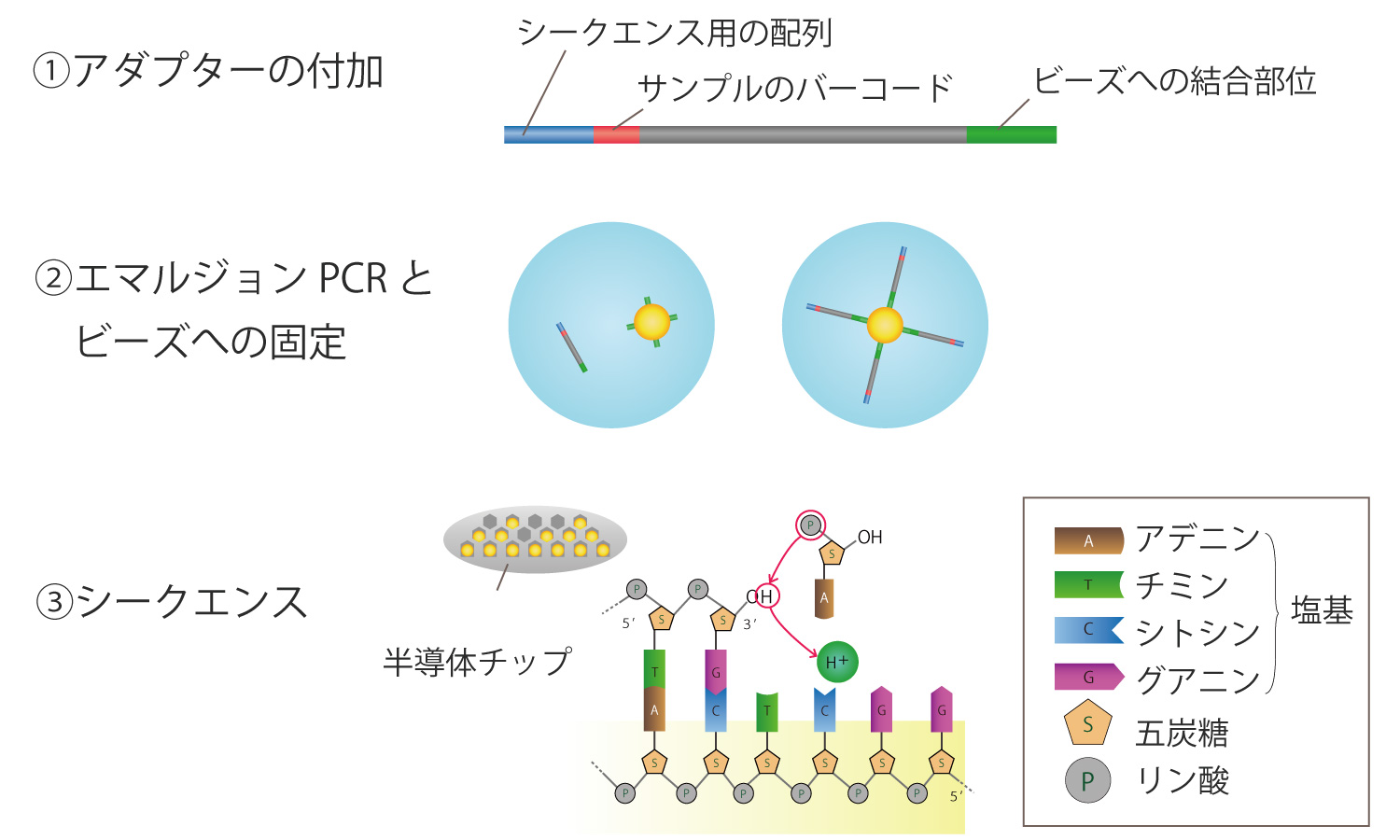
Thermofisher社のIon Torrent system
エマルジョンPCRとDNA伸長によるシークエンス方法です。
数百bpのDNA断片にサンプルが特定できるバーコードとビーズへの結合部位、シークエンス配列を含んだアダプターを付加します。
エマルジョンPCRによってをDNA増幅します。ビーズにアダプターと相補的なオリゴヌクレオチドが付加されており、DNAとビーズを油水エマルジョンで包み込み、エマルジョン内でPCR反応を行います。
シークエンスは半導体チップを用いて行われ、水素イオン濃度を測定することで塩基を特定します。半導体チップには無数の穴が開いており、1つの穴には1つビーズが入ります。dNTPを1種類ずつ送液し、DNA伸長で発生する水素イオン濃度を測定します。
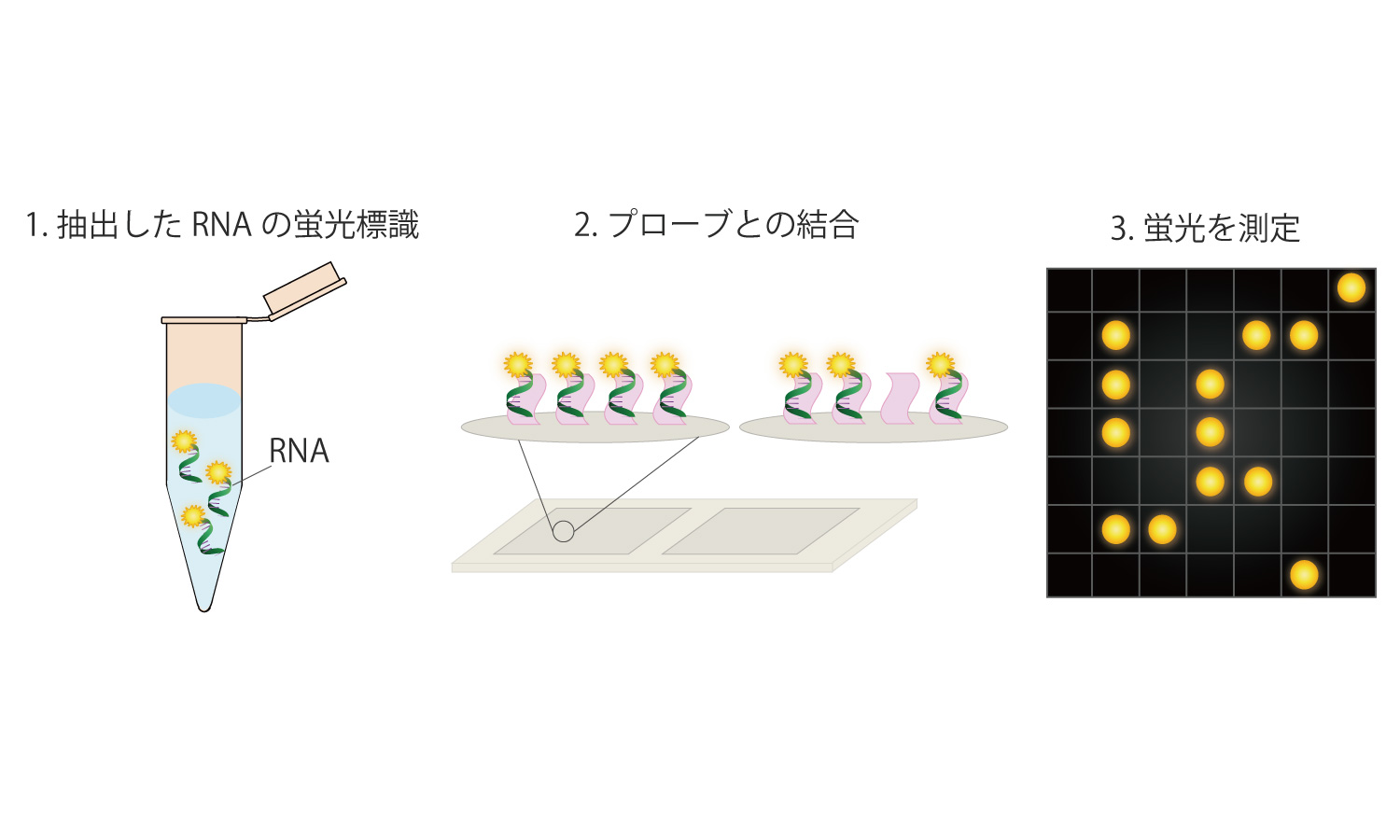
マイクロアレイ
対象物を検出できるプローブを固定化した基盤を使用する方法です。対象物の発現を網羅的に解析することが可能です。
例としてRNAを検出することで病気の有無を判定するマイクロアレイを紹介します。対象者のRNAを抽出し蛍光標識します。基盤には病気の人を区別できるプローブを固定しておき、結合したRNAを蛍光で測定しパターンを見ることで病気の有無を判定します。